Evo-2: ИИ модель для генерации генома, которая знает все древо жизни
История Evo 2
Позвольте мне кратко представить Arc Institute.

Агс Research Institute — это независимый некоммерческий научно-исследовательский институт, расположенный в Калифорнии. Его основная цель заключается в ускорении научного прогресса и изучении фундаментальных причин сложных заболеваний. Институт использует инновационную модель исследований, которая предоставляет ученым полную свободу для изучения направлений, основанных на их любознательности, и одновременно способствует глубокому междисциплинарному сотрудничеству.

Институт Arc и двое ученых, возможно, не так широко известны, но вы наверняка слышали о романтической истории, которая стала популярной в академических кругах в 2022 году. Жених-миллиардер частной исследовательницы из престижного университета сделал ей щедрое пожертвование в размере 500 миллионов долларов. Это позволило ей избежать хлопот, связанных с подачей заявок на проекты, и нанять 150 ученых, чтобы полностью сосредоточиться на научных исследованиях.
Эту женщину-инженера зовут Сильвана Конерманн. Она имеет докторскую степень в области нейробиологии от Массачусетского технологического института (MIT) и ранее работала в лаборатории известного специалиста по CRISPR Фэн Чжана.
Патрик Коллисон — жених-миллиардер (сооснователь Arc Institute) и один из самых молодых миллиардеров в мире, самостоятельно заработавших свое состояние. В 20 лет он бросил учебу в Массачусетском технологическом институте и основал технологическую компанию Stripe, одним из первых инвесторов которой стал Илон Маск. Сегодня в Stripe работают тысячи сотрудников по всему миру.

Патрик Сю, один из основателей Arc Institute, получил степень бакалавра по молекулярной и клеточной биологии в Калифорнийском университете в Беркли в 2010 году. Затем он продолжил обучение [1] в Гарвардском университете, где получил степень магистра по биологии и докторскую степень по биохимии, завершив докторантуру всего за один год. Будучи первым аспирантом Чжан Фэна, он внес значительный вклад в ранние исследования и разработки технологии CRISPR-Cas9.

В декабре 2021 года Патрик Сю вместе с друзьями Патриком Коллисоном и Сильваной Конерманн основали Arc Institute. Это хорошо финансируемое учреждение, которое ценит научную свободу, создало нечто вроде Evo 2.
Кстати, Evo 2 — это улучшенная версия Evo 1. Обучающие данные Evo 1 содержат только данные генома одной клетки. Результаты Evo 1, опубликованные в 2024 году, можно найти в заглавной статье журнала Science.
Введение и мотивация
Прогресс в области методов секвенирования и редактирования ДНК в течение последних десятилетий трансформировал анализ геномных данных в один из фундаментальных инструментов современной биологии. Однако для комплексного анализа последовательностей ДНК, включающего предсказание функциональных эффектов мутаций и рациональное проектирование новых биологических систем, необходимо разработать высокоэффективные методы машинного представления данных. В настоящей работе представлена модель Evo 2 — крупномасштабная языковая модель (Large Language Model), обученная на корпусе из 9 триллионов токенов геномных последовательностей, охватывающих все домены жизни (бактерии, археи, эукариоты, вирусы бактериофагов и прочие).
Концептуальная основа исследования заключается в применении принципов авторегрессионных языковых моделей (по аналогии с методами обработки естественного языка) к символам нуклеотидного алфавита: аденин (A), цитозин (C), гуанин (G) и тимин (T). Результаты демонстрируют, что модель Evo 2 с оптимизированной масштабной архитектурой и репрезентативным набором данных способна выявлять фундаментальные статистические закономерности в последовательностях ДНК, что позволяет эффективно решать следующие задачи:
-
Предсказание функциональной значимости генетических вариантов (Variant Effect Prediction, VEP)
-
Генерация реалистичных геномных последовательностей с возможностью масштабирования до уровня полного генома
-
Оптимизация эпигеномных паттернов, включая моделирование локусов с повышенной доступностью хроматина
Данные и процесс обучения
-
Корпус данных (OpenGenome2): Фундаментальной основой для обучения модели послужил открытый репозиторий OpenGenome2, интегрирующий разнообразные типы последовательностей ДНК (бактериальные и эукариотические геномы, метагеномные данные, последовательности органелл, матричные РНК [2], некодирующие РНК и другие геномные элементы). Совокупный объем корпуса составляет более 9 триллионов нуклеотидов.
-
Параметрическое масштабирование: Разработаны две версии модели Evo 2, дифференцированные по количеству параметров — 7 миллиардов и 40 миллиардов соответственно. Архитектурная оптимизация позволила достичь возможности обработки контекстуального окна размером до 1 миллиона пар оснований (base pairs, bp).
-
Стратифицированная методология обучения:
-
Предварительное обучение (Pretraining): Начальная фаза с ограниченным контекстным окном (8–16 тысяч пар оснований) для идентификации локальных геномных особенностей, включая кодирующие области, регуляторные элементы и функциональные мотивы.
-
Промежуточное обучение (Midtraining): Инкрементальное расширение контекстного окна до 1 миллиона пар оснований, обеспечивающее идентификацию дальнодействующих зависимостей и макроструктурных геномных элементов, таких как оперонная организация у бактерий или сложные интрон-экзонные архитектуры эукариотических генов.
-
-
Архитектура StripedHyena 2: В отличие от традиционных моделей на основе Transformers, архитектура Evo 2 реализована на базе оптимизированного гибридного свёрточно-аттенционного механизма StripedHyena 2, демонстрирующего повышенную вычислительную эффективность при масштабировании контекстного окна.
-
Алгоритмы коррекции репрезентативности (Repeat Down Weighting): Имплементирована система дифференциального взвешивания повторяющихся геномных элементов (тандемных повторов, дупликаций) и интеграция филогенетических маркеров для обеспечения таксономической стратификации последовательностей при обучении модели.
Архитектурные особенности Evo 2, методология обучения и структура датасета
В рамках настоящего исследования разработана инновационная модель Evo 2, обеспечивающая высокоэффективное моделирование геномных последовательностей всех таксономических доменов. Схематическое представление, иллюстрируемое на Рисунке 1, демонстрирует интегральную архитектуру модели, стратифицированные этапы обучения и структуру используемых наборов данных.
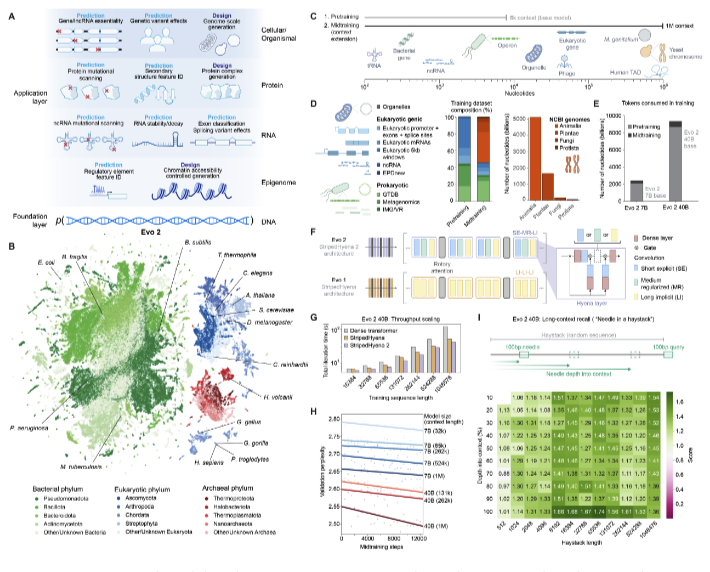
Извиняюсь за качество рисунков, это лучшее, что есть, даже в самой статье именно такое качество.
Как показано на Рисунке 1А, модель Evo 2 концептуализируется как комплексная система интерпретации «нуклеотидного языка», применимая для решения задач различного масштаба: от молекулярного до организменного уровня. Архитектура модели оптимизирована для идентификации структурных особенностей кодирующих и некодирующих регионов, моделирования протеин-нуклеиновых взаимодействий, а также анализа высокоуровневых геномных и эпигеномных паттернов.
Рисунок 1B представляет проекцию геномных последовательностей из обучающего корпуса в двумерное пространство с использованием алгоритма UMAP. Дискретные кластеры точек соответствуют различным доменам и таксономическим группам организмов, дифференцированным по цвету в соответствии с их филогенетической классификацией. Данная визуализация наглядно демонстрирует гетерогенность обучающего корпуса и обосновывает необходимость разработки универсальной модели, способной к эффективной генерализации для всех доменов жизни.
Рисунки 1C–D иллюстрируют бифазную стратегию обучения:
-
Предварительная фаза (pretraining), фокусирующаяся на локальных последовательностных закономерностях и крупных функциональных элементах.
-
Промежуточная фаза (midtraining), характеризующаяся инкрементальным увеличением контекстуального окна до 1 миллиона пар оснований для идентификации глобальных геномных паттернов.
В процессе обучения имплементирован комплекс специализированных методологий, включая дифференциальное взвешивание повторяющихся элементов и оптимизацию батч-распределения данных, направленных на повышение качественных характеристик обучения при работе с протяженными последовательностями и обеспечение репрезентативного покрытия различных функциональных доменов генома.
Рисунок 1E демонстрирует статистическое распределение токенов, использованных на каждой стадии обучения для моделей Evo 2 с 40 и 7 миллиардами параметров соответственно. Очевидно превалирование объема данных на этапе промежуточного обучения с расширенным контекстуальным окном, что обеспечивает оптимальную аппроксимацию к реальным геномным масштабам.
Рисунок 1F представляет схематическую визуализацию архитектуры StripedHyena 2, имплементированной в Evo 2, с тремя дифференцированными типами блоков (SE, MR и LI), структурированными для максимизации эффективности свёрточных и аттенционных операций на масштабных входных последовательностях. Рисунок 1G демонстрирует сравнительный анализ производительности StripedHyena 2 относительно предшествующих версий (StripedHyena 1) и классического Transformer при обучении на 1024 GPU, с очевидным превосходством новой архитектуры в аспекте вычислительной эффективности.
Рисунок 1H отображает результаты валидации модели при вариабельных параметрах длины контекста и масштаба (количества параметров), иллюстрируя положительную корреляцию между расширением контекстуального окна, увеличением параметрического объема модели и улучшением финальных метрик (включая снижение perplexity). В заключение, Рисунок 1I содержит методологическое описание модифицированной задачи «иголка в стоге сена» — специализированного теста на способность Evo 2 к извлечению релевантной информации из контекста протяженностью до 1 миллиона пар оснований. Полученные результаты подтверждают эффективную способность модели к ретенции информации в экстенсивном контексте, что является критически важным параметром при работе с полногеномными последовательностями.
Таким образом, схематическое представление на Рисунке 1 интегрирует ключевые аспекты архитектуры, структуры данных и методологии обучения Evo 2, демонстрируя универсальную применимость модели на всех уровнях биологической организации — от индивидуальных генов до полногеномных последовательностей.
Предсказание эффектов мутаций (Variant Effect Prediction, VEP)
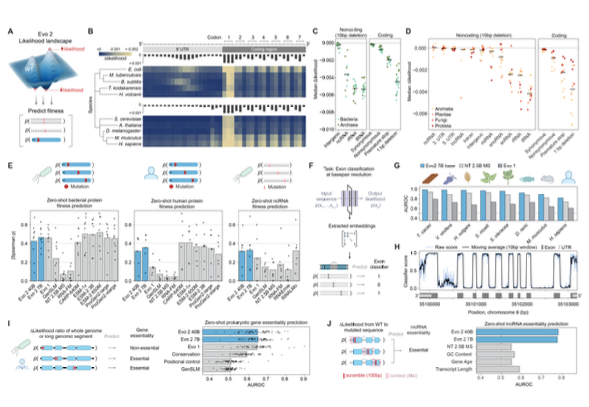
Одной из фундаментальных задач современной геномики является прогнозирование функциональных последствий (патогенных или нейтральных) генетических вариаций. Модель Evo 2 реализует подход без предварительного дообучения (zero-shot), основанный на количественной оценке отношения правдоподобия между мутантной и референсной последовательностями (Рисунок 2A).
Анализ патогенных вариантов ClinVar
Модель демонстрирует превосходную точность в идентификации патогенности мутаций, включая некодирующие варианты и структурные перестройки (инсерции/делеции), превосходя существующие инструменты в режиме без предварительного дообучения. Для специализированных аналитических задач авторы также разработали компактные классификаторы на основе векторных представлений Evo 2, достигающие передовых результатов в прогнозировании вариантов генов BRCA1 и BRCA2, ассоциированных с онкологическими заболеваниями.
Как демонстрирует Рисунок 2B, модель способна эффективно идентифицировать функционально значимые сайты в геномах различных организмов. Анализ изменений в вероятности последовательности при введении мутаций вдоль сайтов начала генов для разнообразных модельных видов выявил, что модель корректно предсказывает сниженную вероятность мутаций в критических элементах: стартовых кодонах белок-кодирующих генов, первых двух основаниях каждого кодона кодирующей области и сайтах связывания рибосом в 5′-нетранслируемой области (5’UTR). Это свидетельствует о способности модели выявлять функционально-консервативные элементы генома в отсутствие эксплицитной аннотации.
Универсальность предсказаний для различных доменов жизни
Рисунки 2C-D демонстрируют результаты стратифицированного анализа вероятности различных типов мутаций в функциональных геномных элементах для прокариотических (2C) и эукариотических (2D) последовательностей с применением версии Evo 2 с 7 миллиардами параметров. Полученные результаты демонстрируют консистентность с фундаментальными биологическими принципами: регионы генома, находящиеся под интенсивным эволюционным давлением, проявляют повышенную чувствительность к мутациям в оценках модели. Медианное изменение вероятности при транзиции от дикого типа к мутантной последовательности визуализировано с дифференциацией по таксономическому домену (для прокариот) или царству (для эукариот), что подтверждает универсальную применимость модели к разнообразным филогенетическим линиям.
Анализ вариаций сплайсинга (SpliceVarDB)
Модель Evo 2 эффективно дифференцирует варианты, нарушающие процесс сплайсинга, в экзонных и интронных областях. Примечательно, что в отличие от традиционных методов предсказания эффектов вариантов, Evo 2 не требует множественных выравниваний последовательностей, но при этом обеспечивает высокую прогностическую точность.
Для валидации прогностической эффективности модели Evo 2 авторы провели комплексный анализ корреляции Спирмена между zero-shot предсказаниями вероятности последовательностей и экспериментальными данными, полученными методом глубокого мутационного сканирования (Deep Mutational Scanning, DMS) для широкого спектра белков и РНК (Рисунок 2E). Эта методология позволяет экспериментально определить функциональные эффекты тысяч вариантов одновременно, что обеспечивает надежную эмпирическую валидацию точности модельных предсказаний.
Дополнительно, авторы имплементировали инновационный классификатор экзонов с однонуклеотидным разрешением, основанный на векторных представлениях (эмбеддингах) Evo 2 (Рисунок 2F). Сравнительный анализ производительности классификаторов, обученных на эмбеддингах различных моделей (Evo 2, Nucleotide Transformer и Evo 1), на выборке из восьми филогенетически дистанцированных видов продемонстрировал превосходство Evo 2 по метрике площади под кривой ROC (AUROC) при идентификации экзонных нуклеотидов (Рисунок 2G). Визуализация предсказаний классификатора для человеческого локуса STOML2 (Рисунок 2H) наглядно демонстрирует точность определения экзон-интронных границ, где вертикальная ось репрезентирует количественную оценку классификатора, а горизонтальная — геномную позицию.
Анализ некодирующих регионов и предиктивная генетика
В дополнение к однонуклеотидным вариантам (SNV) в белок-кодирующих последовательностях, модель демонстрирует высокую эффективность в прогнозировании патогенности вариантов в некодирующих и сплайс-регуляторных элементах, а также в других функциональных геномных компонентах.
Особый интерес [3] представляет применение Evo 2 для предиктивного анализа эссенциальности генов. На Рисунке 2I представлены результаты использования мутационной вероятности вставки преждевременных стоп-кодонов в качестве генетической пертурбации для предикции эссенциальности/неэссенциальности генов у бактерий и бактериофагов. Предсказания модели демонстрируют высокую конкордантность с результатами экспериментальных исследований генной эссенциальности, что подтверждает практическую применимость Evo 2 в функциональной геномике и синтетической биологии.
Аналогичный методологический подход был применен для предикции функциональной значимости длинных некодирующих РНК (lncRNA) человека (Рисунок 2J). Модель эффективно дифференцирует эссенциальные (N = 46) и неэссенциальные (N = 5,417) lncRNA на основе оценки вероятности их случайной реаранжировки (скремблирования последовательности), что было подтверждено результатами экспериментальных скринингов клеточной эссенциальности lncRNA во всех тестируемых линиях.
Таким образом, интегративный анализ предсказательной эффективности Evo 2 в дифференцированных экспериментальных парадигмах подтверждает универсальную применимость модели для прогнозирования функциональных эффектов генетических вариаций во всех таксономических доменах без необходимости дополнительного обучения или параметрической настройки для специфических биологических контекстов.
Генерация геномных последовательностей
В отличие от узкоспециализированных аналитических инструментов, Evo 2 представляет собой генеративную модель, функционирующую на основе авторегрессионного принципа формирования нуклеотидных последовательностей. Экспериментальная валидация модели включала генерацию нескольких типов геномных последовательностей:
-
Митохондриальные геномы человека: Генерация полномасштабных последовательностей протяженностью приблизительно 16 тысяч пар оснований, с корректной реконструкцией функциональных элементов, включая транспортные РНК (tRNA), рибосомальные РНК (rRNA) и консервативные белок-кодирующие гены. Сгенерированные последовательности демонстрируют высокую степень консервации геномной организации (синтении) при одновременной вариабельности на уровне аминокислотных последовательностей.
-
Геном модельного микроорганизма Mycoplasma genitalium: Благодаря расширенному контекстуальному окну (до 1 миллиона пар оснований) модель Evo 2 способна экстраполировать и реконструировать протяженные последовательности (порядка 580 тысяч пар оснований). Сгенерированные геномные последовательности характеризуются сохранением структурной гомологии с известными белковыми доменами (подтверждено анализом с использованием базы данных Pfam) при одновременной дивергенции на уровне первичной структуры, что открывает потенциальные возможности для идентификации новых функциональных вариантов белков.
-
Хромосомы дрожжей Saccharomyces cerevisiae: Продемонстрирована успешная генерация протяженных геномных фрагментов порядка сотен тысяч пар оснований, с корректной реконструкцией функциональных элементов, включая транспортные РНК, интрон-экзонные структуры и регуляторные промоторные области.
Оптимизация эпигеномных паттернов
К категории более сложных аналитических задач относится генерация последовательностей, соответствующих заданным критериям по эпигеномным характеристикам, в частности, локальной доступности хроматина. Для реализации данного подхода авторы имплементировали методологию поиска в процессе инференса (inference-time search):
-
Интеграция предикторных моделей (Enformer, Borzoi): Данные вспомогательные модели осуществляют количественное прогнозирование степени доступности хроматина (открытой конформации) для связывания регуляторных факторов в специфических клеточных типах на основе нуклеотидной последовательности.
-
Алгоритм лучевого поиска (beam search): Процесс генерации осуществляется инкрементально по фрагментам длиной 128 пар оснований, при этом модель Evo 2 генерирует множественные варианты кандидатных последовательностей, а Enformer/Borzoi проводят аналитическую оценку соответствующих эпигеномных профилей. В процессе селективного отбора сохраняются только последовательности, демонстрирующие максимальную конвергенцию с заданным эталонным паттерном, что обеспечивает поэтапное формирование целевой последовательности требуемой протяженности.
-
Экспериментальная демонстрация: Исследователи, моделируя дифференциальные паттерны открытых и компактизированных хроматиновых регионов, продемонстрировали возможность кодирования информационных паттернов, аналогичных коду Морзе (система точек и тире), в виде дискретных эпигеномных пиков.
Существенно отметить, что качественные характеристики генерируемых паттернов демонстрируют позитивную корреляцию с увеличением вычислительных ресурсов (расширение пула кандидатных последовательностей на каждом этапе генерации). Данный феномен иллюстрирует принцип масштабирования вычислительных ресурсов в процессе инференса (inference-time scaling) применительно к биологическим моделям.
Интерпретация внутренних представлений (Sparse Autoencoders)
Учитывая возрастающую сложность и масштабируемость языковых моделей, авторы исследования акцентировали внимание [4] на механистической интерпретируемости. Для этой цели были разработаны и обучены разреженные автоэнкодеры (Sparse Autoencoders, SAE) на основе анализа внутренних активаций Evo 2, с целью идентификации латентных признаков (features), демонстрирующих прямую корреляцию с функциональными биологическими элементами:
-
Регуляторные элементы генной экспрессии: Фильтры разреженных автоэнкодеров эффективно идентифицируют специфические последовательностные мотивы, включая элементы корового промотора (TATA-бокс) и сайты связывания транскрипционных факторов.
-
Структурные элементы белков: В активационных паттернах выявляются характерные признаки, соответствующие основным элементам вторичной структуры белков (α-спирали и β-листы).
-
Профаговые интеграционные сайты и мобильные генетические элементы: Модель демонстрирует способность к автоматической аннотации фаговых инсерций, сайтов CRISPR-спейсеров и других мобильных элементов без эксплицитного обучения на маркированных данных.
-
Структурная организация генов: Определенные латентные признаки коррелируют с экзон-интронной архитектурой, что позволяет идентифицировать структурные компоненты даже в сложноорганизованных генах высших эукариот, включая человека.
Имплементация данных методологических подходов обеспечивает углубленное понимание биологических концептов, выявленных моделью Evo 2 в процессе обучения, и создает основу для высокоточной аннотации неизученных геномов, включая палеогеномные данные (например, геном шерстистого мамонта).
Заключение и перспективы
Представленное исследование позиционирует модель Evo 2 как универсальную платформу для комплексного анализа и рационального дизайна геномных последовательностей. Ключевые результаты исследования могут быть суммированы следующим образом:
-
Таксономическая универсальность: Модель демонстрирует эффективную генерализацию на все три домена жизни, экстраполируя методологию языкового моделирования на структурно и функционально сложные эукариотические геномы.
-
Прогнозирование эффектов вариантов без предварительного обучения (Zero-shot VEP): Evo 2 демонстрирует превосходство над существующими алгоритмами в аспекте прогнозирования функциональных последствий некодирующих и структурных вариантов, функционируя как метод, независимый от множественных выравниваний последовательностей (alignment-free).
-
Полногеномная генерация: Авторы впервые продемонстрировали авторегрессионный подход к реконструкции митохондриальных, бактериальных и эукариотических геномов с интеграцией фрагментов в протяженные контексты порядка сотен тысяч пар оснований.
-
Проектирование эпигеномных ландшафтов: Интеграция вспомогательных предикторных моделей обеспечивает динамическую оптимизацию генерируемых последовательностей в соответствии с заданными эпигеномными паттернами.
-
Доступность и репродуцируемость: Репозиторий данных (OpenGenome2), программный код и параметрические модели (7 и 40 миллиардов параметров) находятся в открытом доступе, что позиционирует Evo 2 как один из наиболее масштабных открытых проектов на пересечении искусственного интеллекта [5] и молекулярной биологии.
Таким образом, модель Evo 2 устанавливает новый методологический стандарт в области фундаментальных биологических моделей, демонстрируя потенциал крупномасштабных языковых моделей в эффективном анализе и моделировании нуклеотидных последовательностей с высокой степенью детализации (от мутационного анализа до геномной архитектуры) на уровне целых хромосом. Перспективные направления развития включают оптимизацию методологий управления инференсом (inference-time steering), интеграцию пространственных 3D-структур хроматина и синергию с экспериментальными методами геномного редактирования, что создаст основу для трансляции парадигмы генеративной биологии в практические приложения — от молекулярной диагностики до синтетической биологии.
🔥Не пропустите важные обновления и углубленные материалы!🔥
Хотите быть в курсе самых свежих обзоров и исследований в мире ML и AI? Переходите по ссылкам ниже, чтобы получить доступ к эксклюзивному контенту:
📌 Все обзоры также доступны в нашем Telegram канале TheWeeklyBrief [6]📢
📌 Более подробный обзор с математической формализацией и программным кодом ждет вас в нашем репозитории Weekly-arXiv-ML-AI-Research-Review [7] 👩💻📂✨
Не упустите шанс глубже погрузиться в мир технологий! 🚀
Автор: Verbasik
Источник [8]
Сайт-источник BrainTools: https://www.braintools.ru
Путь до страницы источника: https://www.braintools.ru/article/12953
URLs in this post:
[1] обучение: http://www.braintools.ru/article/5125
[2] РНК: http://www.braintools.ru/article/8114
[3] интерес: http://www.braintools.ru/article/4220
[4] внимание: http://www.braintools.ru/article/7595
[5] интеллекта: http://www.braintools.ru/article/7605
[6] TheWeeklyBrief: https://t.me/TheWeeklyBrief
[7] Weekly-arXiv-ML-AI-Research-Review: https://github.com/Verbasik/Weekly-arXiv-ML-AI-Research-Review/tree/main/2025/week-10
[8] Источник: https://habr.com/ru/articles/889304/?utm_campaign=889304&utm_source=habrahabr&utm_medium=rss
Нажмите здесь для печати.